
 |
|||||||
|
|
|||||||
|
|
| トップページ > 研究支援実績 > 利用実施例 > 「DNAマイクロアレイ」解析について |
研究支援実績 |
|
マイクロアレイについて |
「DNAマイクロアレイ」解析について |
|
| 「DNAマイクロアレイ」というのは遺伝子の発現量の比較をする実験のための技術です。ではその「遺伝子の発現量比較」とは一体何でしょう。 |
 マイクロアレイスキャナ(左)とスキャンされたマイクロアレイ(右) |
動物・植物・細菌といった生き物は「ゲノム」に書かれた設計図に基づいてできている、ということはご存知のことと思います。この設計図というのは実は沢山の「遺伝子」からできています。いろいろ例外はありますが大まかに言ってこの「遺伝子」は「タンパク質」という生体分子の設計図です。タンパク質というのは食事で大事な栄養素の名前でもありますが、生き物の体を作り、働かせているさまざまな分子のひとつのグループの総称でもあります。たとえば腸で働く消化酵素や病原菌の侵入を防ぐ免疫分子、においを感じるための受容体などもタンパク質の仲間です。タンパク質はアミノ酸という物質(調味料に使われるグルタミン酸、睡眠を改善するトクホとして売られているグリシンなどが有名なアミノ酸です。)がそれぞれのタンパク質によって決まった並び方で互いに結合してできています。そして遺伝子はこのアミノ酸の並び方を記録してある設計図なのです。その記録する素材として使われているのがDNA(デオキシリボ核酸)です。このDNAは砂糖の仲間であるデオキシリボースという糖、アデニン(A)、グアニン(G)、シトシン(C)、チミン(T)という4種類のうちどれか一つの塩基、それにリン酸という3つの要素からできており、ゲノム上の遺伝子はこのDNAが多数連結したものです。どのようにDNAを使ってアミノ酸の並び方を記録しているかというとA、G、C、Tの4種の塩基の並び方で記録しています。3つの塩基の並び方でひとつのアミノ酸を決めるのです。(GGAやGGCなら先ほどのグリシン、GAAかGAGならグルタミン酸、となります。)また、DNAは細胞の中で通常二本のDNA鎖が弱く結合して二重らせんといわれる構造をとっています。(この二重らせんの構造は生命にとってとても重要なことで、この構造を決めたワトソン博士とクリック博士がノーベル賞を受けたのは広く知られています。)二本のDNA鎖は先に挙げた塩基という部分で水素結合という方法で結合しています。ACGTの各塩基それぞれが決まった相手と水素結合を作ります。その組み合わせはAとT、CとG、です。また、DNA鎖には5’→3’という方向があり二重らせんはこの方向が互い逆向きに結合します。片方の鎖がATCGGGTTという並びであるなら二本鎖DNAは下記のようになります。 |
5’ ATCGGGTT 3’ 3’ TAGCCCAA 5’ |
また、このように互いに二重らせんを作って結合できる配列(ATCGGGTTとAACCCGAT)のことを互いに相補的な配列、と呼びます。 |
さて、ゲノム上に書かれている生き物の設計図である遺伝子が実際に働くためには、設計図に書かれている機械が実際に役立つには実際の機械を作らなければならないのとまったく同様にそこに書かれているタンパク質をつくらなければなりません。実際の工場では原本の設計図からコピーをとり、そのコピーを使うことがあるように、DNAでできているゲノム上の設計図はメッセンジャーRNA(mRNA)という分子にコピーされ、このmRNAを用いてタンパク質がつくられます。RNAというのはリボ核酸というものでDNAに大変よく似ています。DNAの糖の部分、デオキシリボースがRNAではリボースという糖になっていますがRNAもDNA同様、糖、塩基、リン酸という3つの要素からできています。今一つ、RNAでの塩基はアデニン(A)、グアニン(G)、シトシン(C)はDNAと共通ですがチミン(T)のかわりにウラシル(U)という塩基が使われています。mRNAは細胞内で一本鎖で存在しておりゲノムDNAのように二本鎖で存在しているわけではありません。しかしDNA同様、相補的な配列を持つRNAあるいはDNAと結合し、二本鎖になることはできます。 さて、設計図である遺伝子は一つの細胞に哺乳類などの場合は父型と母型の2つがあり、これはその細胞でその遺伝子からタンパク質がたくさんつくられようとほとんどつくられまいと同じ数です。しかし設計図のコピーであるmRNAは細胞の中でたくさんつくられているタンパク質についてはたくさんつくられることが多いのでこのmRNAが細胞中にどのくらいあるかを調べることでどのタンパク質がどのくらいつくられているかの見当をつけることができます。 どのタンパク質がどの細胞・組織でつくられているか、あるいはあまりつくられていないか、を知ることは生物を調べるためだけでなく医療などの応用の面からも大変重要です。例えば細胞ががん化していく際には普段は少ししかつくられていなかったがん関連遺伝子からのタンパク質が多めにつくられる、というようなことがあります。そこで近年よく行われる研究のしかたとして病気になってしまった組織・細胞と健康な組織・細胞それぞれでどのようなタンパク質がつくられているか比較する、ということがあります。ただ、組織・細胞のなかのタンパク質をすべて捕まえてきて調べるのは大変困難な仕事です。それはタンパク質にはとても多くつくられるもの(細胞の形を支えるタンパク質など)とごくわずかしかつくられないもの(外界の刺激をつたえる受容体など)の量の差がきわめて大きいばかりでなく、タンパク質の種類を決定することは今でもRNAやDNAに書かれた設計図を解読するよりも困難だからです。そこでタンパク質を全て調べるかわりに設計図のコピーであるmRNAの量を調べることもこのような比較の実験では実施されています。「DNAマイクロアレイ」というのはこのmRNAの量の比較をする実験手法のための技術です。 ではDNAマイクロアレイとはどんなものでどのような仕組みで使われているのかをみてみましょう。 |
DNAマイクロアレイとはなにか |
|
DNAマイクロアレイとは一言で言うと遺伝子の特徴を表すような遺伝子の一部分と同じDNAをスライドガラス上に沢山はりつけたもの、です。 遺伝子は4種類の塩基の並び方で記述されています。つまり違う遺伝子の間ではこの塩基の並び方が違うのです。この塩基の並び方のことを塩基の配列(塩基配列)と呼びます。また多くの遺伝子が数百から数千個の塩基の配列でできています。ヒトのもので2万から3万種類近くあるといわれている遺伝子のすべての塩基配列を全長で取り扱うことは困難です。そこで各遺伝子の塩基配列からそれぞれの遺伝子の特徴となる塩基配列をなるべく短く取り出してくることができれば全長を扱う必要はなくなります。 4種類の塩基がたとえば5つならぶと4x4x4x4x4=1024通りの並び方が可能です。この順列の考え方で単純に3万以上の並び方が可能な塩基配列の長さを考えると 4の10乗はおよそ3万2千となりますから最短10塩基の配列で3万種類の区別は可能になります。けれども似た性質を持つ遺伝子同志は塩基配列も似ているので実際に使われているマイクロアレイには20塩基から60塩基くらいの長さの塩基配列が用いられています。なお、これらの各遺伝子の特徴を示す配列をもつDNAのことを各遺伝子のプローブ(探針)と呼びます。DNAマイクロアレイとは特定の場所に特定の遺伝子プローブが張り付けられたものなのです(図1)。 |
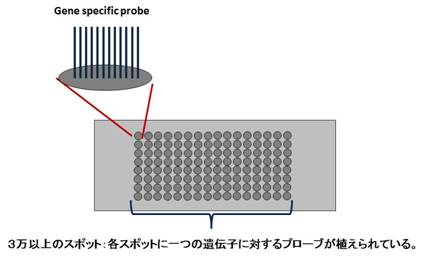 図1:DNAマイクロアレイ |
この遺伝子の特徴を表す塩基配列(プローブ)を基板上に固定化するところにナノテクが使われています。初期には試験管の中で化学的に合成したDNAを針の先につけてガラス板にスポットするなどしてマイクロアレイを作成していましたがこのような方法では一枚のガラス基板上に乗せられる遺伝子数には限りがあり、とてもゲノム上の遺伝子すべてを調べることはできませんでした。 アジレント社ではマイクロアレイを作成するのに表面を加工したガラス板の上にDNAを直接合成する、という方法でスライドガラスの上に16万本以上のプローブを固定化することに成功し、ゲノム配列から考えうるすべての遺伝子配列を一枚のスライドガラス上で調べることができるようになりました。これは大変精密なインクジェットプリンターを用いて狙った位置に決まった試薬を打ち込み、DNAを合成していく、というものです。 またアフィメトリクス社では光リソグラフィーというICを作成する技術を用いて基板上で狙った塩基配列を高密度に位置を指定して合成する方法を確立し、やはり一枚のチップの上でゲノム上のすべての遺伝子の解析を行うことを可能にしています。 さて、このように高密度に全遺伝子に対するプローブを持ったマイクロアレイで実際に遺伝子発現を解析するにはDNAまたはRNAが互いに相補的な塩基配列に結合することを利用します。簡単に言ってしまえばある細胞からmRNAを取出し、マイクロアレイに乗せ、各遺伝子のプローブにmRNAがどれだけ結合したかを量ればよいのです(図2)。 |
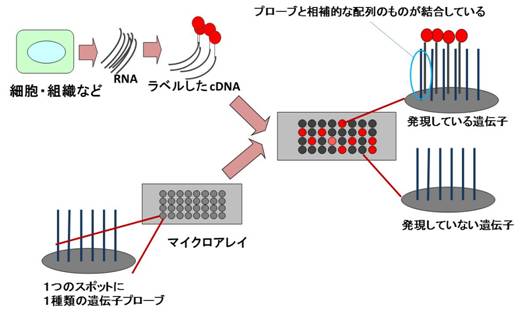 図2:マイクロアレイ解析(文献1より改変して引用) |
結合量をはかるのにほとんどの場合で蛍光を利用します。マイクロアレイ上の各遺伝子プローブの位置で蛍光の強さを測定し、その蛍光の量からmRNAの結合量を推定します。その方法には大きく分けて2つの方法があります。ひとつは一色法でこれは一つの対象からとったRNAに蛍光試薬を反応させ、各mRNAが蛍光を発するようにし、マイクロアレイにのせます(図3)。 |
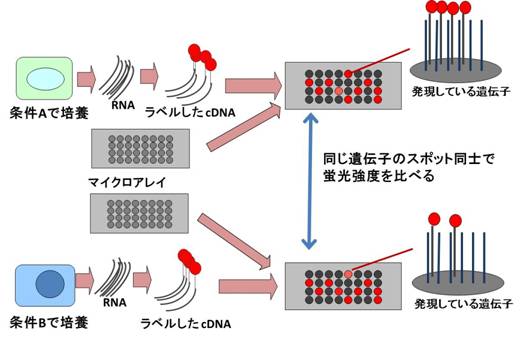 図3:マイクロアレイ解析 一色法(文献1より改変して引用) |
もう一つの方法は二色法でこれは発現量を比較したい2つの細胞、組織からそれぞれmRNAをとり、それぞれ違う色の蛍光試薬を反応させます。これらをほぼ同量になるように混ぜ合わせて一枚のマイクロアレイにのせ、2色の蛍光それぞれの強度を測定し、その比をとります。比をとることで発現量の変化を追跡します(図4)。 |
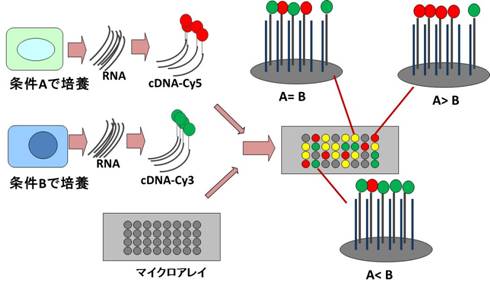 図4:マイクロアレイ解析 二色法(文献1より改変して引用) |
このような方法で2つのサンプルの間の遺伝子発現量を比較することのできるマイクロアレイは、例えば病気になっている組織となっていない組織の間での発現量の変化を解析し、病気とかかわりの深い遺伝子を探す、ということに用いられています。 マイクロアレイは現在さまざまな研究・開発に用いられており、マイクロアレイを使った解析結果のデータベースとしてはアメリカ合衆国の国立衛生研究所のNCBIにGEO、日本のDDBJにCIBEXというデータベースが構築されています。 現在ではゲノム解析が盛んになり、遺伝子の塩基配列を読み取る能力が大幅に進歩し、配列解析のコストが下がり、マイクロアレイではなく遺伝子の配列解析を用いて、組織・細胞に発現している遺伝子を全て(網羅的に)突き止めてしまう、という方法もとられるようになってきました。しかしながら網羅的な配列解析では解析して出てきた配列すべてをどの遺伝子であるか同定したり、それぞれの数を数えたり、という配列データ取得後の労力が大きいことや、いくらコストが下がったといってもまだまだマイクロアレイのほうが安価であることなどから遺伝子発現量の解析にはまだ当分はマイクロアレイが重要な手段として使われていくことになるでしょう。 |
文献 |
|
1. Hanagata N., Takemura T. and Minowa T.: Global gene expression analysis for evaluation and design of biomaterials. |
|
||||||||||||||||